Table des matières
Les CATIs 3G
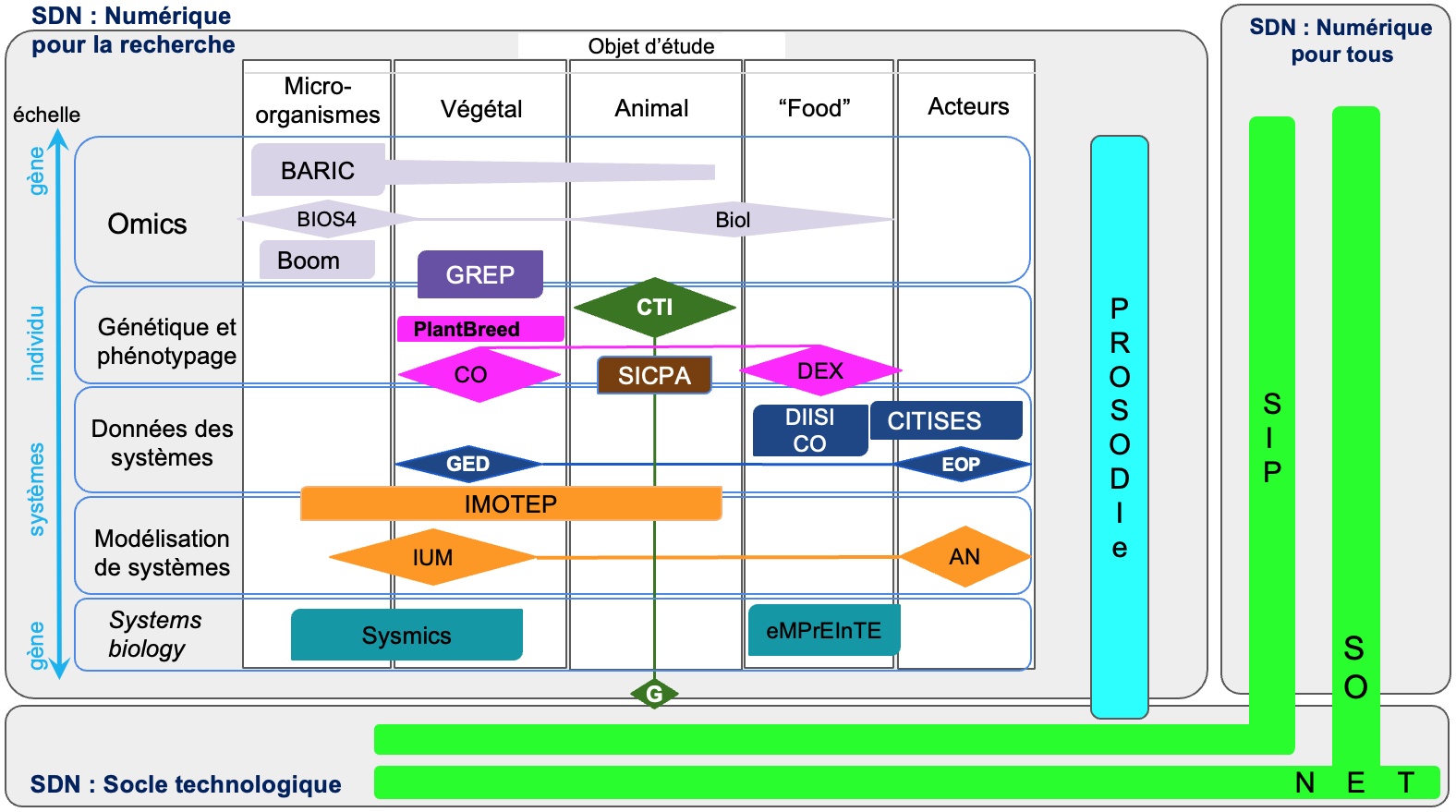
Vue d'ensemble des propositions de CATI-3G à la phase d'AMI. L'objectif était de situer ces propositions par rapport aux domaines du SDN. Pour le domaine de recherche, nous avons tenté d’aller plus loin en les situant dans un espace croisant 3 dimensions : l’“objet” d’étude, les échelles étudiées (des gènes aux agro-socio-écosystèmes et l’activité dominante sur un gradient données–modèles. L’objectif était aussi de voir les adhérences, interactions, chevauchements possibles à ces propositions. Pour cela, nous avons aussi proposé des clusters : Omics, Génétique et phénotypage, données des systèmes, modélisation des systèmes et “systems biology”. La figure composée de 2 losanges reliés par un trait représente des CATIs se situant sur plusieurs objets voire domaines non voisins sur le graphique.
Liste des CATIs
| Acronyme | Nom Détaillé Porteur(s) Opérationnels(s) | Département(s)/DAR pilote(s) | Effectif 2019 - 2025 |
|---|
| BARIC | Bioinformatique pour l’Analyse, la Représentation et Intégration de Connaissances | Martine DA ROCHA, Nicolas LAPALU & Fabrice LEGEAI (SPE) | [SPE], BAP, AGROECOSYSTEM | 30—>33 |
| BIOS4Biol | Bio-informatique et Statistiques pour la Biologie / Bioinformatics Statistics for Biology | Luc JOUNEAU (SA), Géraldine PASCAL (PHASE) | [MATHNUM , GA], ECODIV, BAP, TRANSFORM, PHASE, SA | 34—>49 |
| BOOM | Bioinformatics for Omics and metaOmics of Microbes | Hélène CHIAPELLO (MICA), Valentin LOUX (MATHNUM) | [MATHNUM, MICA], PHASE | 29—>36 |
| CITISES | Centre Informatisé du Traitement de l'Information en Sciences Économiques et Sociales | Annie HOFSTETTER, Fabrice LEVERT (ECOSOCIO), Alban THOMAS (ECOSOCIO) | [ECOSOCIO], ALIMH | 34—>35 |
| CODEX | COnnaissance et Données EXpérimentales | Eric LATRILLE (AGROECOSYSTEM), Isabelle ALIC (MATHNUM) | [MATHNUM, AGROECOSYSTEM], TRANSFORM, SPE, BAP, MICA, ALIMH | 22—>39 |
| CTIG | Centre de Traitement de l’Information Génétique | Marc LAVAL (GA) | [GA] | 16—>12 |
| DIISCICO | Développements Informatiques pour l’Instrumentation, le Suivi, le Contrôle et l’Intégration des Connaissances sur les prOcédés | Patrice BUCHE, Christophe LANGRUME (SPE) | TRANSFORM, MATHNUM, ACT, ALIMH, PHASE, ECODIV, AGROECOSYSTEM, SPE | 41—>30 |
| eMPrEInTE | Molecular PhEnotyping and bIochemical daTa Engineering | Franck GIACOMONI (ALIMH), David LEGLAND (TRANSFORM) | [ALIMH, TRANSFORM] | 17—>36 |
| GEDEOP | GEstion des Données d’Expérimentations, d’Observations et de Pratiques sur les agro-socio-éco-systèmes | Wilfried HEINTZ (ECODIV), Amélie FIOCCA (DipSO) | [ECODIV], ACT, AGROECOSYSTEM, AQUA, MATHNUM, SPE | 66—>93 |
| GREP | Genomics Research Environment for Plants | Johann CONFAIS & Raphaël FLORES (BAP) | [BAP], SPE, ECODIV | 26—>20 |
| IMOTEP | Information, Modèles et Traitement des données en Epidémiologie et dynamique de Populations | Thierry HOCH (SA), Emily WALKER (MATHNUM) | MATHNUM, SA, SPE, ECODIV, ALIMH | 31—>43 |
| IUMAN | Informatisation et Utilisation des Modèles pour les Agroécosystèmes Numériques | Patrick CHABRIER (MATHNUM) | [AGROECOSYSTEM, MATHNUM], ACT, ECODIV, PHASE, ECOSOCIO, SPE, AQUA | 51—>55 |
| PlantBreed | Software Environment for Plant Selection | Alain CHARCOSSET, Yannick DE OLIVEIRA & Jacques LAGNEL (BAP) | [BAP] | 18—>23 |
| PROSODIe | Compilations Reproductibilité et Qualité pour l’Informatique Scientifique | Sophie AUBIN & Tovo RABEMANANTSOA (DipSO) | [MATHNUM, DipSO], BAP, DICSDAR, TRANSFORM, ALIMH, ECODIV, AGROECOSYSTEM, AQUA | 19—>32 |
| SICPA | Systèmes d'informations et calcul pour le phénotypage animal | François LAPERRUQUE (GA), Anissa DIEUDONNÉ & Bernadette URBAN (PHASE) | [GA, PHASE] | 28—>41 |
| SIP | Services Informatiques de Proximité | Patrick BERGER (TRANSFORM), Ghyslain SEVENIER (AGROECOSYSTEM) | DSI et départements (ACT, AGROECOSYSTEM,ALIMH…) | ~100—>95 |
| SoNET | Socle numérique et expertises transverses | Olivier SCHNEIDER (DSI) | DSI, Diagonal, DRH, DICSDAR, DPCE, DPI, GEVES, DiFA | ~150—>176 |
| SysMics | Systems Biology for Omics | Sandra DÉROZIER (MATHNUM), Johann JOETS (BAP) | [MATHNUM, BAP], MICA | 18—>21 |
La commission d’homologation
Comme pour les deux premières campagnes d’homologation a été mise, une commission a été mise en place en 2017. L’objectif était notamment que cette troisième génération de Catis soit en mesure de servir les nouveaux enjeux du numérique. La commission a été construite avec la volonté d’incorporer des niveaux de compétences, de points de vue, et de responsabilités diversifiées dans les domaines de l’informatique scientifique et d’appui.
Sa composition était la suivante :
- Laurent Bruckler, PC Occitanie-Montpellier, Président de la commission (participation à la commission Catis 2G 2012)
- Michaël Chelle, Délégué scientifique à la Transition numérique
- Christophe Caron, unité Ingenum, en charge du volet e-infrastructure
- Hélène Chiapello, en charge des Pepis (et du lien avec Cati), de la formation et GPEC dans l’unité Ingenum, puis ingénieure dans l’unité MIA MaIAGE
- Frédéric Toppan, unité Ingenum, Gestionnaire d’unité
- Thierry Caquet, CD EFPA puis directeur scientifique environnement (responsable scientifique du CATI SIOEA, impliqué dans les CATIq CGI et IUMA)
- Hervé Monod, CD MIA (implication dans plusieurs Cati, notamment thématiques « omiques », modélisation et calcul)
- Sylvie Dequin, CD MICA (implication dans le Cati MIAGO)
- Stéphane Ingrand CDA PHASE (responsable scientifique du CATI SICPA)
- Jean Michel Perez-Cano, Directeur des Systèmes d’information (responsable du Cati ICARE) – puis relais de Françoise Roudaut –
- Sylvie Ladet, unité Dynafor département SAD (responsable opérationnel du Cati Action depuis 2009).
- Anne Goelzer, unité MaIAGE, département MIA (responsable opérationnel du Cati MIAGO)
Les étapes pour l’homologation
Les principales étapes de travail de la commission ont été les suivantes :
- 26 avril 2017 : 1re journée de travail de la commission, Paris (bilan CATIs 2012)
- 11 Juillet 2017 : 2e journée de travail de la commission, Paris (principes de construction des CATIs-3G)
- 3 Octobre 2017 : Réunion avec les Chefs de départements sur les CATIs
- 9 Octobre 2017 : Assemblée générale « Inter CATIs-2G » et ateliers de travail pilotés par la commission CATIs-3G
- 29 Janvier 2018 : 3e journée de travail de la commission, Paris (phasage avec le SDN)
- 30 Mas 2018 : Journée de présentation et d’échanges de la commission avec les porteurs potentiels de CATIs-3G et annonce de lancement de l’appel à manifestation d’intérêt (AMI)
- Mai 2018 : Réception des réponses à l’appel à manifestation d’intérêt pour les CATIs-3G
- 31 Mai : 4e journée de travail de la commission, Paris (analyse des réponses à l’appel à manifestation d’intérêt)
- Juin 2018 : Diffusion de la synthèse et des préconisations de la commission sur les appels à manifestation d’intérêt pour les CATIs-3G
- Septembre 2018 : Réception des dossiers d’homologation pour les CATIs-3G
- 3 Octobre 2018 : 5e journée de travail de la commission, Paris (analyse des dossiers d’homologation)
- Octobre 2018 : Synthèse et recommandations sur les dossiers d’homologation pour les CATIs-3G
La commission d’homologation a opéré en 2 phases, l’appel à manifestation d’intérêt et l’homologation des projets finaux. La première phase a abouti à 21 manifestations d’intérêt. A l’issue de l’analyse de celles-ci, la commission a proposé d’aller vers des regroupements, fusions, et reconfigurations plus fortes et les plus cohérentes possibles de plusieurs projets de CATIs, aboutissant potentiellement à des CATIs de plus grande taille partageant des questions transversales communes. Pour la seconde phase, la commission d’homologation a reçu 18 projets finaux, soit un peu moins qu’en 2012 (21 CATIs avaient été homologués, dont 18 scientifiques). Un travail « en amont », conduit par les départements (et dans bien des cas, en inter départements) et par les directions d’appui, en lien avec les Catis, a été nécessaire pour construire des CATIs ayant un réel adossement à une orientation scientifique ou d’appui assumé.
Les documents à télécharger
- Analyse de la commission d'homologation CATI-3G sur les AMI : Avis.pdf
- Ensemble des dossiers de réponse à l'AMI : AMI2018_CATI-3G
- Ensemble des dossiers de demande d'homologation : CATI-3G_dossiers
- Avis définitif de la commission d'homologation : Synthese_et_analyse_sur_les_dossiers_complets_pour_les_CATI_3G.pdf